

使用 Kmeans聚类实现颜色的分割
source link: https://blog.51cto.com/domi/5568873
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.
之前分享过kmeans算法(传送门: 数据挖掘算法—K-Means算法),这期分享一下使用 Kmeans聚类实现颜色的分割,使用 L*a*b* 颜色空间和 K 均值聚类自动分割颜色。
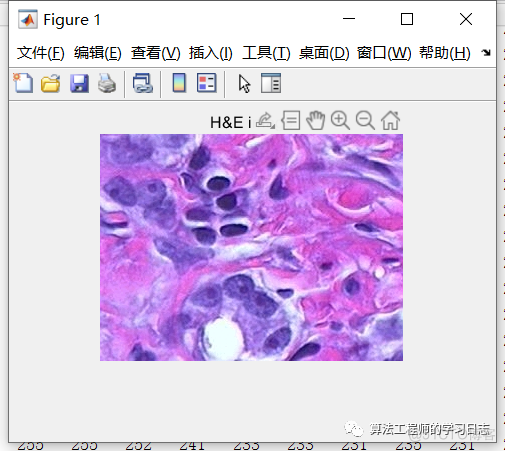
步骤 1:读取图像
读取hestain.png,
步骤 2:将图像从 RGB 颜色空间转换为 L*a*b* 颜色空间
L*a*b* 颜色空间(也称为 CIELAB 或 CIE L*a*b*)能够量化视觉差异。
L*a*b* 颜色空间是从 CIE XYZ 三色值派生的。L*a*b* 空间包含光度层 'L*'、色度层 'a*'(表示颜色落在沿红-绿轴的位置)和色度层 'b*'(表示颜色落在沿蓝-黄轴的位置)。所有颜色信息都在 'a*' 和 'b*' 层。可以使用欧几里德距离度量来测量两种颜色之间的差异。
使用 rgb2lab 将图像转换为 L*a*b* 颜色空间。
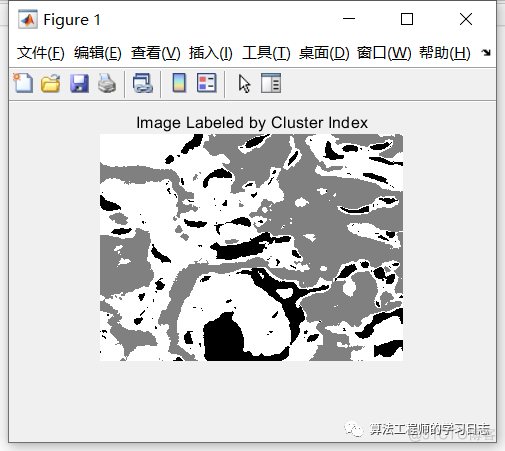
步骤 3:用 K 均值聚类对基于 'a*b*' 空间的颜色进行分类
聚类是一种分离对象组的方法。K 均值聚类将每个对象视为在空间中有一个位置。它将对象划分为若干分区,使每个簇中的对象尽可能彼此靠近,并尽可能远离其他簇中的对象。K 均值聚类要求您指定要划分的簇数和用于量化两个对象之间距离的距离度量。
由于颜色信息基于 'a*b*' 颜色空间,因此您的对象是具有 'a*' 和 'b*' 值的像素。将数据转换为数据类型 single,以便与 imsegkmeans 结合使用。使用 imsegkmeans 对对象进行聚类以分为三个簇。
对于输入中的每个对象,imsegkmeans 会返回一个对应于簇的索引或标签。用像素的标签标注图像中的每个像素。
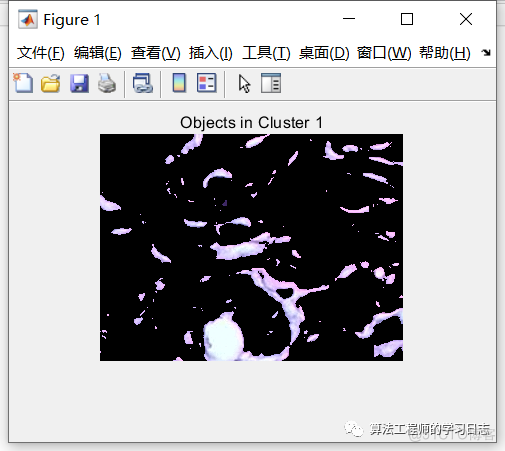
步骤 4:创建按颜色分割 H&E 图像的图像
使用 pixel_labels,可以按颜色分离 hestain.png 中的对象,这将产生三个图像。
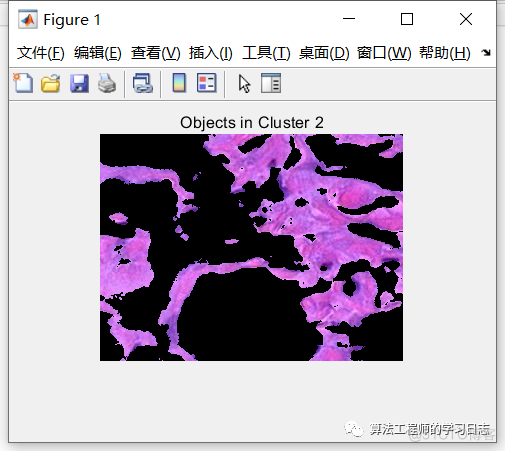
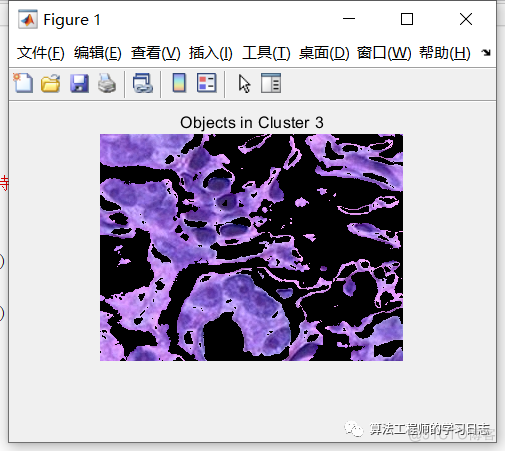
步骤 5:分割核
簇 3 包含蓝色对象。请注意,有深蓝色和浅蓝色对象。您可以使用 L*a*b* 颜色空间中的 'L*' 层来分离深蓝色和浅蓝色。细胞核为深蓝色。
前面提到过,'L*' 层包含每种颜色的亮度值。提取此簇中像素的亮度值,并使用 imbinarize 用全局阈值对其设置阈值。掩膜 is_light_blue 给出了浅蓝色像素的索引。
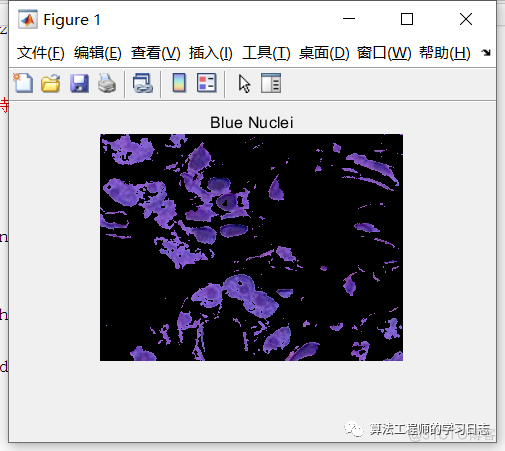
复制蓝色对象的掩膜 mask3,然后从掩膜中删除浅蓝色像素。将新掩膜应用于原始图像并显示结果。只有深蓝色细胞核可见。
blue_nuclei = he .* uint8(mask_dark_blue);imshow(blue_nuclei)title('Blue Nuclei');
Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK