

首个大众可用PyTorch版AlphaFold2复现,哥大开源,star量破千
source link: https://www.51cto.com/article/712278.html
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.
首个大众可用PyTorch版AlphaFold2复现,哥大开源,star量破千-51CTO.COM
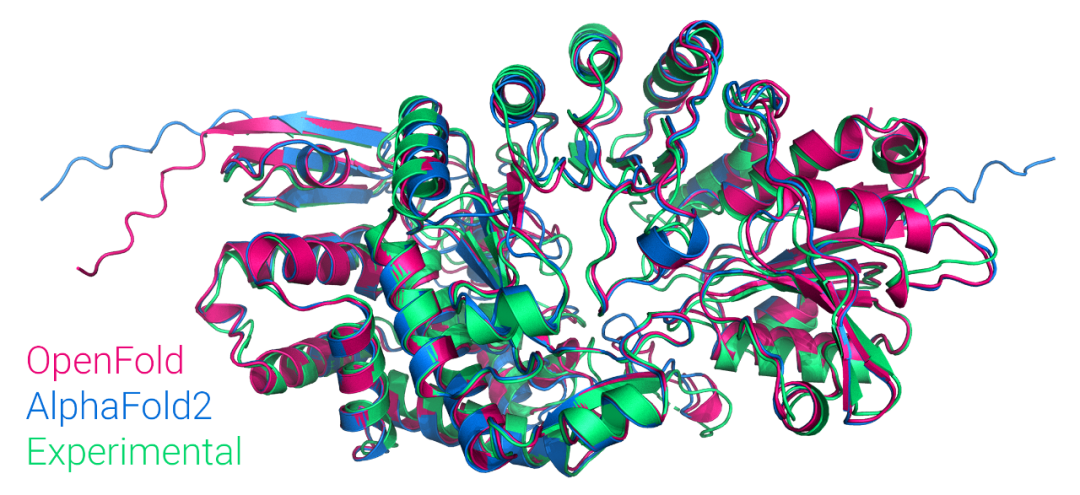
刚刚,哥伦比亚大学系统生物学助理教授 Mohammed AlQuraishi 在推特上宣布,他们从头训练了一个名为 OpenFold 的模型,该模型是 AlphaFold2 的可训练 PyTorch 复现版本。Mohammed AlQuraishi 还表示,这是第一个大众可用的 AlphaFold2 复现。

AlphaFold2 可以周期性地以原子精度预测蛋白质结构,在技术上利用多序列对齐和深度学习算法设计,并结合关于蛋白质结构的物理和生物学知识提升了预测效果。它实现了 2/3 蛋白质结构预测的卓越成绩并在去年登上了《自然》杂志。更令人惊喜的是,DeepMind 团队不仅开源了模型,还将 AlphaFold2 预测数据做成了免费开放的数据集。
然而,开源并不意味着能用、好用。其实,AlphaFold2 软件系统的部署难度极大,并且对硬件的要求高、数据集下载周期长、占用空间大,每一条都让普通开发者望而却步。因此,开源社区一直在努力实现 AlphaFold2 的可用版本。
这次哥伦比亚大学 Mohammed AlQuraishi 教授等人实现的 OpenFold 总训练时间大约为 100000 A100 小时,但在大约 3000 小时内就达到了 90% 的准确率。

OpenFold 与原版 AlphaFold2 的准确率相当,甚至略胜一筹,可能因为 OpenFold 的训练集更大一点:
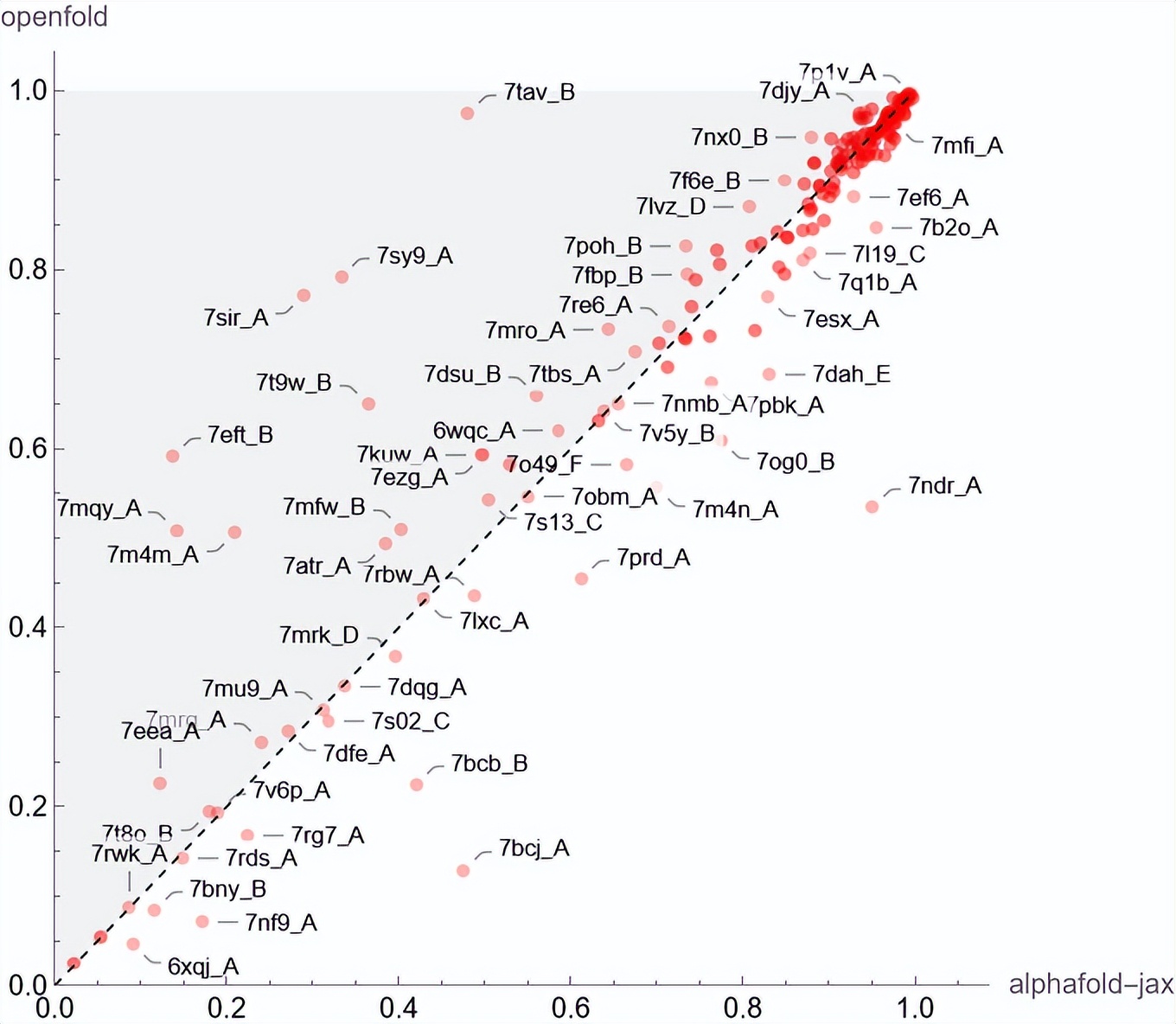
OpenFold 的主要优势是推理速度显著提升,对于较短的蛋白质序列,OpenFold 的推理速度可以达到 AlphaFold2 的两倍。另外,由于使用自定义的 CUDA 内核,OpenFold 使用更少的内存就能推理更长的蛋白质序列。
OpenFold 介绍
OpenFold 几乎再现了原始开源推理代码 (v2.0.1) 的所有功能,除了已趋于被淘汰的「模型集成」功能,该功能在 DeepMind 自己的消融测试中就表现不佳。
无论是否有 DeepSpeed,OpenFold 都能以全精度或 bfloat16 进行训练。为了实现 AlphaFold2 的原始性能,该团队从头开始训练 OpenFold,现已公开发布了模型权重和训练数据。其中,训练数据包含大约 400000 份 MSA 和 PDB70 模板文件。OpenFold 还支持使用 AlphaFold 的官方参数进行蛋白质推理。
与其他实现相比,OpenFold 具有以下优点:
- 短序列推理:加快了在 GPU 上推理少于 1500 个氨基酸残基的链的速度;
- 长序列推理:通过该研究实现的低记忆注意力(low-memory attention)对极长链进行推理,OpenFold 可以在单个 A100 上预测 超过 4000 个残基的序列结构,借助 CPU offload 甚至可以预测更长的序列;
- 内存高效在训练和推理期间,在 FastFold 内核基础上修改的自定义 CUDA 注意力内核,使用的 GPU 内存分别比等效的 FastFold 和现有的 PyTorch 实现少 4 倍和 5 倍;
- 高效对齐脚本:该团队使用原始 AlphaFold HHblits/JackHMMER pipeline 或带有 MMseqs2 的 ColabFold,已经生成了数百万个对齐。
Linux 系统下的安装与使用
开发团队提供了一个在本地安装 Miniconda、创建 conda 虚拟环境、安装所有 Python 依赖项并下载有用资源的脚本,包括两组模型参数。
运行以下命令:
scripts/install_third_party_dependencies.sh使用如下命令激活环境:
source scripts/activate_conda_env.sh停用命令:
source scripts/deactivate_conda_env.sh在激活环境下,编译 OpenFold 的 CUDA 内核
python3 setup.py install在 / usr/bin 路径下安装 HH-suite:
# scripts/install_hh_suite.sh使用如下命令可以下载用于训练 OpenFold 和 AlphaFold 的数据库:
bash scripts/download_data.sh data/如果要使用一组 DeepMind 的预训练参数对一个或多个序列进行推理,可以运行如下代码:
python3 run_pretrained_openfold.py \
fasta_dir \
data/pdb_mmcif/mmcif_files/ \
--uniref90_database_path data/uniref90/uniref90.fasta \
--mgnify_database_path data/mgnify/mgy_clusters_2018_12.fa \
--pdb70_database_path data/pdb70/pdb70 \
--uniclust30_database_path data/uniclust30/uniclust30_2018_08/uniclust30_2018_08 \
--output_dir ./ \
--bfd_database_path data/bfd/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt \
--model_device "cuda:0" \
--jackhmmer_binary_path lib/conda/envs/openfold_venv/bin/jackhmmer \
--hhblits_binary_path lib/conda/envs/openfold_venv/bin/hhblits \
--hhsearch_binary_path lib/conda/envs/openfold_venv/bin/hhsearch \
--kalign_binary_path lib/conda/envs/openfold_venv/bin/kalign
--config_preset "model_1_ptm"
--openfold_checkpoint_path openfold/resources/openfold_params/finetuning_2_ptm.pt更多细节请参见 GitHub:https://github.com/aqlaboratory/openfold
扩展阅读:
- 高效预测几乎所有人类蛋白质结构,AlphaFold 再登 Nature,数据库全部免费开放
- 生物计算专家超细致解读 AlphaFold2 论文:模型架构及应用
- DeepMind 开源的 AlphaFold 怎么用?打开 Colab 就能在线用
Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK