

复杂网络社区发现算法python实战准备.
source link: https://shartoo.github.io/2020/02/11/network-community-python-pratice/
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.

1 要安装的包
# 不要单独安装networkx和community ,会导致Graph没有best_parition属性
# 安装与networkx 2.x 版本对应的python-louvain(它内部包含了community)
pip install -U git+https://github.com/taynaud/python-louvain.git@networkx2
# 安装 networkx,理论上应该默认安装最新版版的 2.4
pip install networkx
# 如果上述安装之后,'Graph' object has no attribute 'edges_iter'
#需要卸载networkx 2.x版本,只能使用1.x版本。注意networkx1.x版本的函数API 不一样,
pip install networkx==1.9.1
2 数据和参考资料
3 测试代码
我们下载使用 astro-gh数据集。
import community
from community import community_louvain
data = "./data/astro-ph/astro-ph.gml"
Graph=nx.read_gml(data)
# network2.x的图划分
part = community_louvain.best_partition(Graph)
# network1.x的图划分
part =community.best_partition(Graph)
测试代码2
import community
import networkx as nx
import matplotlib.pyplot as plt
#better with karate_graph() as defined in networkx example.
#erdos renyi don't have true community structure
#G = nx.erdos_renyi_graph(30, 0.05)
data = "./data/astro-ph/astro-ph.gml"
G=nx.read_gml(data)
#first compute the best partition
partition = community.best_partition(G)
#drawing
size = float(len(set(partition.values())))
pos = nx.spring_layout(G)
count = 0.
for com in set(partition.values()) :
count = count + 1.
list_nodes = [nodes for nodes in partition.keys()
if partition[nodes] == com]
nx.draw_networkx_nodes(G, pos, list_nodes, node_size = 20,
node_color = str(count / size))
nx.draw_networkx_edges(G, pos, alpha=0.5)
plt.show()
运行时间有点长,大概需要10分钟,效果如下
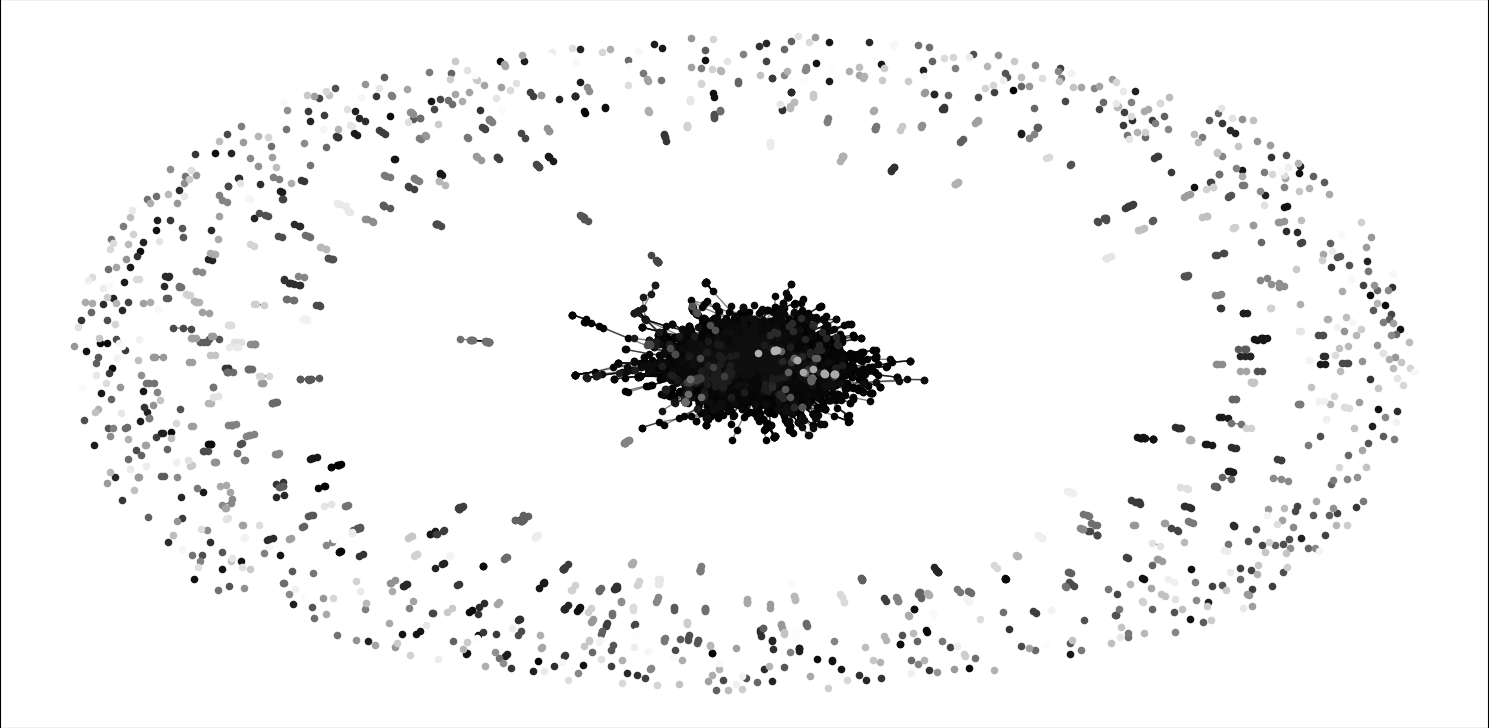
4 相关API
4.1 community
community API 函数比较少,主要是从网络中划分社区,而社区划分算法又只有一个默认实现。网络部分由另外一个包networkx实现。
community.best_partition(graph, partition=None, weight='weight', resolution=1.0, randomize=None, random_state=None):使用Louvain heuristices方法划分的获得最高模块度的社区发现算法。graph:networkx.Graph:需要划分的网络图。一般由直接读取的数据得到networkx.read_gml(data)partition:dict, optional:里面是词典形式的数据,k为节点id,value为节点的标签。算法将以此为基础开始运算得到新的划分。weight:str, optional:字符串形式的权重。作为权重的运算resolution:double, optional: 改变社区的规模尺寸,默认为1.描述的是论文Laplacian Dynamics and Multiscale Modular Structure in Networks中所说的规模。randomize:boolean, optional: 是否随机节点和社区的评估顺序,来获得不同的划分结果random_state:int,optional:上面随机数的随机种子返回结果:新的词典形式的划分结果,key为节点id或名字,value为新的所属社区id(从0开始增长)
community.generate_dendrogram(graph, part_init=None, weight='weight', resolution=1.0, randomize=None, random_state=None):以层次图的形式划分社区。树状图中每一层都是图中节点的一个划分。第0层为第一个划分,包含了最小的社区,最佳社区长度是树状图层次减去1,层次越高社区规模越大。graph:networkx.Graph;需要划分的网络图part_init:dict, optional: 算法起始社区划分,词典形式,key为节点,value为节点对应的社区- 其他参数与上面一样
import community
from community import community_louvain
import networkx as nx
G=nx.erdos_renyi_graph(50, 0.1)
dendo =community_louvain.generate_dendrogram(G)
for level in range(len(dendo) - 1) :
print("partition at level", level,"is", community_louvain.partition_at_level(dendo, level))
partition at level 0 is {0: 0, 1: 1, 2: 2, 3: 3, 4: 4, 5: 5, 6: 5, 7: 6, 8: 7, 9: 3, 10: 8, 11: 5, 12: 9, 13: 10, 14: 11, 15: 12, 16: 10, 17: 11, 18: 3, 19: 11, 20: 8, 21: 10, 22: 13, 23: 7, 24: 6, 25: 14, 26: 13, 27: 2, 28: 0, 29: 13, 30: 13, 31: 5, 32: 9, 33: 6, 34: 7, 35: 15, 36: 14, 37: 10, 38: 11, 39: 2, 40: 13, 41: 15, 42: 12, 43: 3, 44: 1, 45: 0, 46: 4, 47: 12, 48: 16, 49: 16}
partition at level 1 is {0: 0, 1: 1, 2: 2, 3: 1, 4: 3, 5: 3, 6: 3, 7: 2, 8: 4, 9: 1, 10: 4, 11: 3, 12: 0, 13: 2, 14: 3, 15: 2, 16: 2, 17: 3, 18: 1, 19: 3, 20: 4, 21: 2, 22: 5, 23: 4, 24: 2, 25: 1, 26: 5, 27: 2, 28: 0, 29: 5, 30: 5, 31: 3, 32: 0, 33: 2, 34: 4, 35: 1, 36: 1, 37: 2, 38: 3, 39: 2, 40: 5, 41: 1, 42: 2, 43: 1, 44: 1, 45: 0, 46: 3, 47: 2, 48: 5, 49: 5}
community.partition_at_level(dendrogram, level):返回指定层次的社区划分结果。使用示例如上代码。community.induced_graph(partition, graph, weight='weight'):产生社区聚合图,在社区之间产生一条带权重w的边,如果社区内边的总权重为w的话。- 其他参数跟上面一样
返回值:一个新的图,其中节点为划分。
4.2 networkx
networkx 包括网络(图)的构建,添加/删除节点、边等。使用networkx构建网络图时,节点可以是任意可哈希的对象,边可以与任意对象关联。
- 构建网络图:
G=networkx.Grapgh()即可构建一个空的网络图。网络是由节点和边组成的集合。 - 节点
- 添加一个节点:
G.add_node(1) - 添加节点列表:
G.add_nodes_from([2, 3]) - 使用包含节点的迭代器添加节点:
H=networkx.path_graph(10),G.add_nodes_from(H),此时的图H包含了一些节点,但是被视为新的节点,也可以用G.add_node(H)的方式将H作为一个节点来添加。 - 节点删除。
Graph.remove_node()删除一个节点,Graph.remove_nodes_from()删除多个节点
- 添加一个节点:
- 边:如果边已经存在,再次添加不会报错。
- 可以一次添加一条边。
G.add_edge(1,2) - 也可以一次添加多条边。边列表添加,
G.add_edges_from([(1, 2), (1, 3)])。也可以使用边迭代器G.add_edges_from(H.edges) - 边删除。
Graph.remove_edge(1,3)删除一条边,Graph.remove_edges_from()删除多条边
- 可以一次添加一条边。
- 图的统计属性。我们可以查看的属性有
G.nodes,G.edges,G.adj,G.degree(都是集合形式存放)。效果如下
list(G.nodes)
[1, 2, 3, 'spam', 's', 'p', 'a', 'm']
list(G.edges)
[(1, 2), (1, 3), (3, 'm')]
list(G.adj[1]) # or list(G.neighbors(1))
[2, 3]
G.degree[1] # the number of edges incident to 1
2
- 图的子集的统计属性。上面的四种属性都接收参数以指定特定的节点或边或节点子集。
G.edges([2, 'm'])
EdgeDataView([(2, 1), ('m', 3)])
G.degree([2, 3])
DegreeView({2: 1, 3: 2})
- 其他方式得到节点邻居。比如
Graph[1]就可以得到节点1的所有邻居,Graph[1][2]得到节点1和2之间的边的属性。甚至可以修改属性,Graph[1][2]['color']=""blue等同于Graph.edges[1,2]['color']="blue"。 - 使用
G.adjacency()或G.adj.items()即可访问所有(节点,邻居)对。注意,如果是无向图,每条边可能会出现两次。
FG = nx.Graph()
FG.add_weighted_edges_from([(1, 2, 0.125), (1, 3, 0.75), (2, 4, 1.2), (3, 4, 0.375)])
for n, nbrs in FG.adj.items():
for nbr, eattr in nbrs.items():
wt = eattr['weight']
if wt < 0.5: print('(%d, %d, %.3f)' % (n, nbr, wt))
(1, 2, 0.125)
(2, 1, 0.125)
(3, 4, 0.375)
(4, 3, 0.375)
- 给图,节点,边添加属性,权重、标签、颜色等,可以是任意Python对象
- 给图加/修改属性。
networkx.Graph(day="Friday"),修改G.graph['day'] = "Monday" - 节点属性。
add_node(),add_nodes_from(),G.nodes - 边属性。使用
add_edge()和add_edges_from()
- 给图加/修改属性。
# 节点属性添加和修改
>>> G.add_node(1, time='5pm')
>>> G.add_nodes_from([3], time='2pm')
>>> G.nodes[1]
{'time': '5pm'}
>>> G.nodes[1]['room'] = 714
>>> G.nodes.data()
NodeDataView({1: {'time': '5pm', 'room': 714}, 3: {'time': '2pm'}})
# 边属性修改/添加
>>> G.add_edge(1, 2, weight=4.7 )
>>> G.add_edges_from([(3, 4), (4, 5)], color='red')
>>> G.add_edges_from([(1, 2, {'color': 'blue'}), (2, 3, {'weight': 8})])
>>> G[1][2]['weight'] = 4.7
>>> G.edges[3, 4]['weight'] = 4.2
- 有向图:使用
DG = networkx.DiGraph()可以构建有向图。特有的属性是DiGraph.out_edges(),DiGraph.in_degree(),DiGraph.predecessors(),DiGraph.successors(),有向图的节点度数等于入度和出度之和。有向图的neighbours()等同于successors()。如果你想把有向图转换为无向图可以直接使用Graph.to_undirected()或者networkx.Graph(G)
>>> DG = nx.DiGraph()
>>> DG.add_weighted_edges_from([(1, 2, 0.5), (3, 1, 0.75)])
>>> DG.out_degree(1, weight='weight')
0.5
>>> DG.degree(1, weight='weight')
1.25
>>> list(DG.successors(1))
[2]
>>> list(DG.neighbors(1))
[2]
多边图:如果两个节点之间要存在多条边,可以使用
MG = nx.MultiGraph(),节点1和2之间添加多条不同权重的边的示例MG.add_weighted_edges_from([(1, 2, 0.5), (1, 2, 0.75), (2, 3, 0.5)]),可以使用此图来计算最短路径。图的计算和操作:
- subgraph(G, nbunch):抽取图G的子图,子图中节点由nbunch给出
- union(G1,G2): 图合并
- disjoint_union(G1,G2):假定所有节点都不同,合并两个图
- cartesian_product(G1,G2):计算两个图的笛卡尔积
- compose(G1,G2):合并两个图根据两个图中共有节点
- complement(G):补充图
- create_empty_copy(G):构造一个该图子类的空备份
- to_undirected(G):转化为无向图
- to_directed(G):转化为有向图
生成图
- 各种经典小图数据。如彼得森图
networkx.petersen_graph()会生成一个有固定节点和边的彼得森图 - 也可以按照给定节点数生成其他经典图,如5个节点的完全图
networkx.complete_graph(5),完全二分图nx.complete_bipartite_graph(3, 5) - 随机图生成器:
er = nx.erdos_renyi_graph(100, 0.15),red = nx.random_lobster(100, 0.9, 0.9)等。 - 从数据源格式读取:支持各种图存储格式读取。边列表,邻居列表,GML,GraphML,pickle,LEDA等。
- 各种经典小图数据。如彼得森图
分析图:可以对图做各种分析,比如连通图分析,最短路径分析等。所有支持的计算算法 特别多。。
画图:此包并非用于画图,但是可以用Python matplotlib和Graphviz软件包接口一起画图。
5 使用Python实际分析网络数据
5.1 构建网络
参考教程
载入相关包和数据
import csv
from operator import itemgetter
import networkx as nx
#This part of networkx, for community detection, needs to be imported separately.
from networkx.algorithms import community
# 载入数据,假设节点名称列表和边列表分别位于两个不同的csv文件 quakers_nodelist.csv 和 quakers_edgelist.csv
with open('quakers_nodelist.csv', 'r') as nodecsv:
nodereader = csv.reader(nodecsv)
nodes = [n for n in nodereader][1:]
node_names = [n[0] for n in nodes] # Get a list of only the node names
with open('quakers_edgelist.csv', 'r') as edgecsv:
edgereader = csv.reader(edgecsv)
edges = [tuple(e) for e in edgereader][1:]
创建网络图,并加入节点和边,并输出基本信息
G = nx.Graph()
G.add_nodes_from(nodes)
G.add_edges_from(edges)
print(nx.info(G))
得到网络基本信息
Name:
Type: Graph
Number of nodes: 119
Number of edges: 174
Average degree: 2.9244
假若上述的节点信息列表包含了其他列,比如name,historical significance,gender,birth year,death year,SDFB ID属性,你想将这些属性也加入到网络中。networkx允许给节点和边添加属性nx.set_node_attributes()和nx.set_edge_attributes()。你需要将这些属性转化为词典形式,key为节点名,value为属性值,需要对每个属性创建一个词典。
hist_sig_dict = {}
gender_dict = {}
birth_dict = {}
death_dict = {}
id_dict = {}
循环遍历所有节点,给不同属性赋值
# 一个节点有多列,依次为 name,historical significance,gender,birth year,death year,SDFB ID
for node in nodes: # Loop through the list, one row at a time
hist_sig_dict[node[0]] = node[1]
gender_dict[node[0]] = node[2]
birth_dict[node[0]] = node[3]
death_dict[node[0]] = node[4]
id_dict[node[0]] = node[5]
然后通过set_node_attributes将所有属性词典添加到网络图
nx.set_node_attributes(G, hist_sig_dict, 'historical_significance')
nx.set_node_attributes(G, gender_dict, 'gender')
nx.set_node_attributes(G, birth_dict, 'birth_year')
nx.set_node_attributes(G, death_dict, 'death_year')
nx.set_node_attributes(G, id_dict, 'sdfb_id')
# 测试访问节点的birth_year属性
for n in G.nodes():
print(n, G.nodes[n]['birth_year'])
5.2 网络分析
分析之前我们需要知道要分析的网络数据的大体情况,比如有多少节点,多少边。节点太少(小于15个节点,不如直接画出来)或太多(比如150万个节点,分析子集)都不利于分析。当前无向图网络相对于有向图更容易分析。
5.2.1 网络图外观
网络图外观显示了节点之间如何连接的,因为网络图有拓扑结构,可以看出连接关系,数据分布中心化还是去中心化的,稠密的还是稀疏的,圆形 还是线型连接居多,是聚合在一起还是分散的。当前分析的数据集(Quaker)使用Gephi(Force-directed分布(对于小数据集可以创建干净、易于理解的图))可视化效果如下

可视化能分析到的东西基本到此为止。更详细的分析需要量化。
稠密度(density):一个不错的开始分析指标。所有节点实际边数目除以所有可能连接数。此参数可以让你快速了解网络连接情况。nx.density(G)最短路径分析:相对复杂,并且在大网络可视化时不容易发现。主要适用于找到朋友的朋友,参考六度定律。假若我们要计算节点A到B之间最短路径。nx.shortest_path(G, source="A", target="B"),会输出从A到B节点的中间节点列表。如果某个节点是位于很多最短路径中间,此节点一般是Hub,并且重要性较高。半径:顺着最短路径分析,可以分析很多其他指标。比如半径(diameter),如果直接对当前网络计算半径会出错,因为网络包含很多子图,子图之间不连通。可以分析网络找到最大连通图,然后计算其半径。components = nx.connected_components(G);largest_component = max(components, key=len)。如果最大连通图的半径为8,表明最长的最短路径为8.
三重闭合度(triadic closure):如果两个人都认识同一个人,那么这两个人也可能彼此认识,这会在网络中形成一个三角形。网络中这些闭合三角形的数量可以用于发现网络中聚类簇和社区。nx.transitivity(G)聚类系数clustering coefficient:衡量网络中三角闭合度的一般称为聚类系数,因为它表示的是聚集的倾向。但是结构化的网络图使用的是transitivity代表的是所有存在的三角数量除以所有可能的三角数量的比例。刻画的是网络内部的连接。- 切记,考察如
transitivity和稠密度(density)时多考虑似然度(likehoods)而非certainties。transitivity让你能顺着连接性理解网络,
5.2.2 中心度
下一步是分析网络中哪些节点比较重要。分析节点重要程度的方法有很多。包括degree(度),betweenness centrality(中介性),eigenvector centrality(特征向量中心性)。
有较多度数的节点称之为hub,计算节点度数是找到hub的最快方法。这些hub是网络中关键,比如当前数据集中最高度数的节点是此关系网创建人,其他度数少一些的是共同创建人。
# 计算所有节点的度,然后将其作为网络属性词典
degree_dict = dict(G.degree(G.nodes()))
nx.set_node_attributes(G, degree_dict, 'degree')
print(G.nodes['William Penn'])
# 对所有节点按照度从大到小排序
import itemgetter
# itemgetter(1) 代表要对degree_dict.items中第2个列排序
sorted_degree = sorted(degree_dict.items(), key=itemgetter(1), reverse=True)
eigenvector centrality(特征向量中心性)是度数的一种拓展,结合了节点的边以及节点的邻居。它计算的是如果你是一个hub,以及你与都少个hub连接。其取值范围是0到1,值越大越具有中心性。对于理解哪些节点能迅速传递信息十分有帮助。PageRanke算法是特征向量中心性的拓展。
betweenness centrality(介中性)与另外两种度量方法不同,它不关心某个/些节点的边数目。它关心的是所有通过单个节点的最短路径。为计算这些最短路径,首先得计算网络中所有可能的最短路径,所以此度量计算比较耗时,其取值范围也是0到1。使用它很容易找到网络中分离的两个子网。如果两个聚类簇之间仅存在一个节点,这两个聚类簇之间的所有社区都必须经过此节点。与hub相反,此节点称之为broker。尽管介中性不是发现broker的唯一手段,但是是最有效率的。它让你知道,某些节点虽然与它们相连的节点数少,但是它们是网络子网之间桥梁
betweenness_dict = nx.betweenness_centrality(G) # Run betweenness centrality
eigenvector_dict = nx.eigenvector_centrality(G) # Run eigenvector centrality
# 将属性赋予网络
nx.set_node_attributes(G, betweenness_dict, 'betweenness')
nx.set_node_attributes(G, eigenvector_dict, 'eigenvector')
# 根据介中性 排序
sorted_betweenness = sorted(betweenness_dict.items(), key=itemgetter(1), reverse=True)
print("Top 20 nodes by betweenness centrality:")
for b in sorted_betweenness[:20]:
print(b)
</div
Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK