

典型相关分析介绍及python实现
source link: https://www.bobobk.com/581.html
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.

典型相关分析介绍及python实现
2021年12月29日
在处理单个高维数据时,通过可以通过LDA,PCA,等等方法进行降维处理,但是如果某两个数据来自同一个样本,但是数据类型不同,差距巨大时,怎么办呢?这个时候就是典型相关性分析(Canonical Correlation Analysis,CCA)的应用场景.CCA允许我们同时从两套数据分析.典型的应用场景就包括生物学上的联合分析,同一组样本,同时检测转录组和蛋白组,转录组和代谢组以及微生物代谢组等等,更详细的内容可参考维基百科.
CCA与PCA的联系与差别
CCA有点类似PCA(主成分分析,principal component analysis),它们都由同一个课题组提出,在降维方面(canonical variables)可以认为是多套数据的PCA.
不同之处是PCA旨在找出一套数据中能够表示最多方差的线性组合,而CCA旨在找出两套数据中能够最大程度表示其相关性的线性组合.
python实现CCA分析
那么在python中如何实现CCA分析呢? 在sklearn包中的cross_decomposition提供了CCA分析方法,直接调用即可,这里以企鹅数据为例
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
filename = "penguins.csv"
df = pd.read_csv(filename)
df =df.dropna()
df.head()
species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g sex
0 Adelie Torgersen 39.1 18.7 181.0 3750.0 MALE
1 Adelie Torgersen 39.5 17.4 186.0 3800.0 FEMALE
2 Adelie Torgersen 40.3 18.0 195.0 3250.0 FEMALE
4 Adelie Torgersen 36.7 19.3 193.0 3450.0 FEMALE
5 Adelie Torgersen 39.3 20.6 190.0 3650.0 MALE
我们选取其中两种特征作为研究对象bill_length_mm和bill_depth_mm作为一组,而flipper_length_mm与body_mass_g作为另一组,
from sklearn.preprocessing import StandardScaler
df1 = df[["bill_length_mm","bill_depth_mm"]]
df1_std = pd.DataFrame(StandardScaler().fit(df1).transform(df1), columns = df1.columns)
df1_std.head()
df2 = df[["flipper_length_mm","body_mass_g"]]
df2_std = pd.DataFrame(StandardScaler().fit(df2).transform(df2), columns = df2.columns)
df2_std.head()
# df 1
bill_length_mm bill_depth_mm
0 -0.896042 0.780732
1 -0.822788 0.119584
2 -0.676280 0.424729
3 -1.335566 1.085877
4 -0.859415 1.747026
# df2
flipper_length_mm body_mass_g
0 -1.426752 -0.568475
1 -1.069474 -0.506286
2 -0.426373 -1.190361
3 -0.569284 -0.941606
4 -0.783651 -0.692852
开始正式的CCA分析
from sklearn.cross_decomposition import CCA
ca = CCA()
xc,yc = ca.fit(df1, df2).transform(df1, df2)
其得到的结果与输入长度一致
np.shape(xc)
# (333,2)
np.shape(yc)
# (333,2)
而且是高度相关的,这里直接计算其相关性
np.corrcoef(xc[:, 0], yc[:, 0])
np.corrcoef(xc[:, 1], yc[:, 1])
相关性结果为
array([[1. , 0.78763151],
[0.78763151, 1. ]])
array([[1. , 0.08638695],
[0.08638695, 1. ]])
第一对结果很好,相关性0.79,而第二对结果堪忧,我们将CCA分析得到的结果与物种和性别组合成新的dataframe
cca_res = pd.DataFrame({"CCA1_1":xc[:, 0],
"CCA2_1":yc[:, 0],
"CCA1_2":xc[:, 1],
"CCA2_2":yc[:, 1],
"Species":df.species,
"sex":df.sex})
cca_res.head()
CCA1_1 CCA2_1 CCA1_2 CCA2_2 Species sex
0 -1.186252 -1.408795 -0.010367 0.682866 Adelie MALE
1 -0.709573 -1.053857 -0.456036 0.429879 Adelie FEMALE
2 -0.790732 -0.393550 -0.130809 -0.839620 Adelie FEMALE
4 -1.718663 -0.542888 -0.073623 -0.458571 Adelie FEMALE
5 -1.772295 -0.763548 0.736248 -0.014204 Adelie MALE
通过其第一列数据进行散点图分析
sns.scatterplot(data=cca_res,x="CCA1_1",y="CCA2_1",hue="Species",s=10)
plt.title(f'First column corr = {np.corrcoef(cca_res.CCA1_1,cca_res.CCA2_1)[0, 1]:.2f}')
plt.savefig("cca_first.png",dpi=200)
plt.close()
其图为:
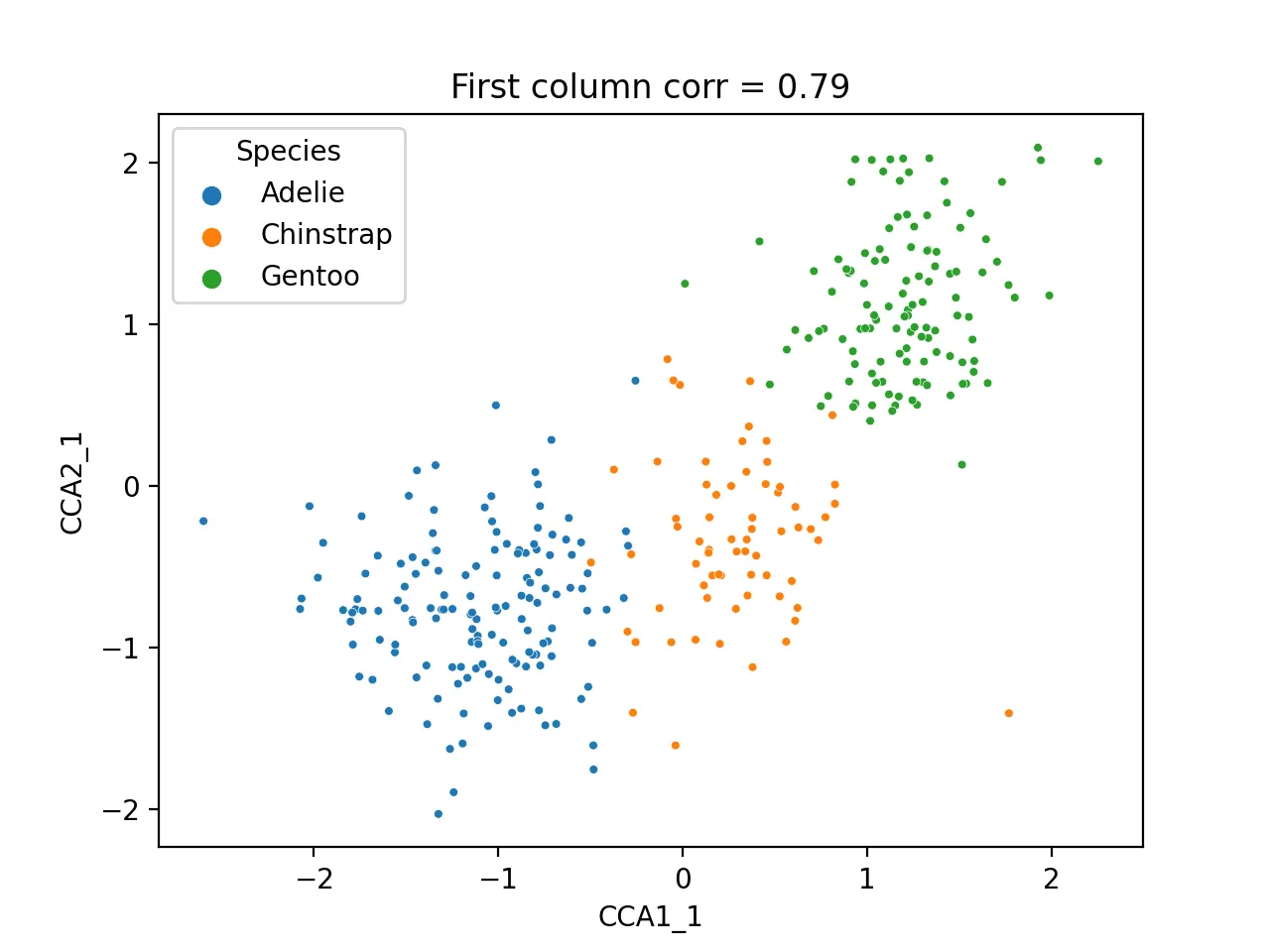 鉴于第二列数据相关性低,这里通过与原始矩阵进行相关性热图比较,找到其可能的体现的数据
鉴于第二列数据相关性低,这里通过与原始矩阵进行相关性热图比较,找到其可能的体现的数据
cca_df = pd.DataFrame({"cca1_1":cca_res.CCA1_1,
"cca1_2":cca_res.CCA1_2,
"cca2_1":cca_res.CCA2_1,
"cca2_2":cca_res.CCA2_2,
"Species":df.species.astype('category').cat.codes,
"Island":df.island.astype('category').cat.codes,
"sex":df.sex.astype('category').cat.codes})
dfcor = cca_df.corr()
mask = np.triu(np.ones_like(dfcor))
sns.heatmap(dfcor,cmap="bwr",annot=True)
plt.savefig("cca_corr.png",dpi=200)
plt.close()
绘制的图为:
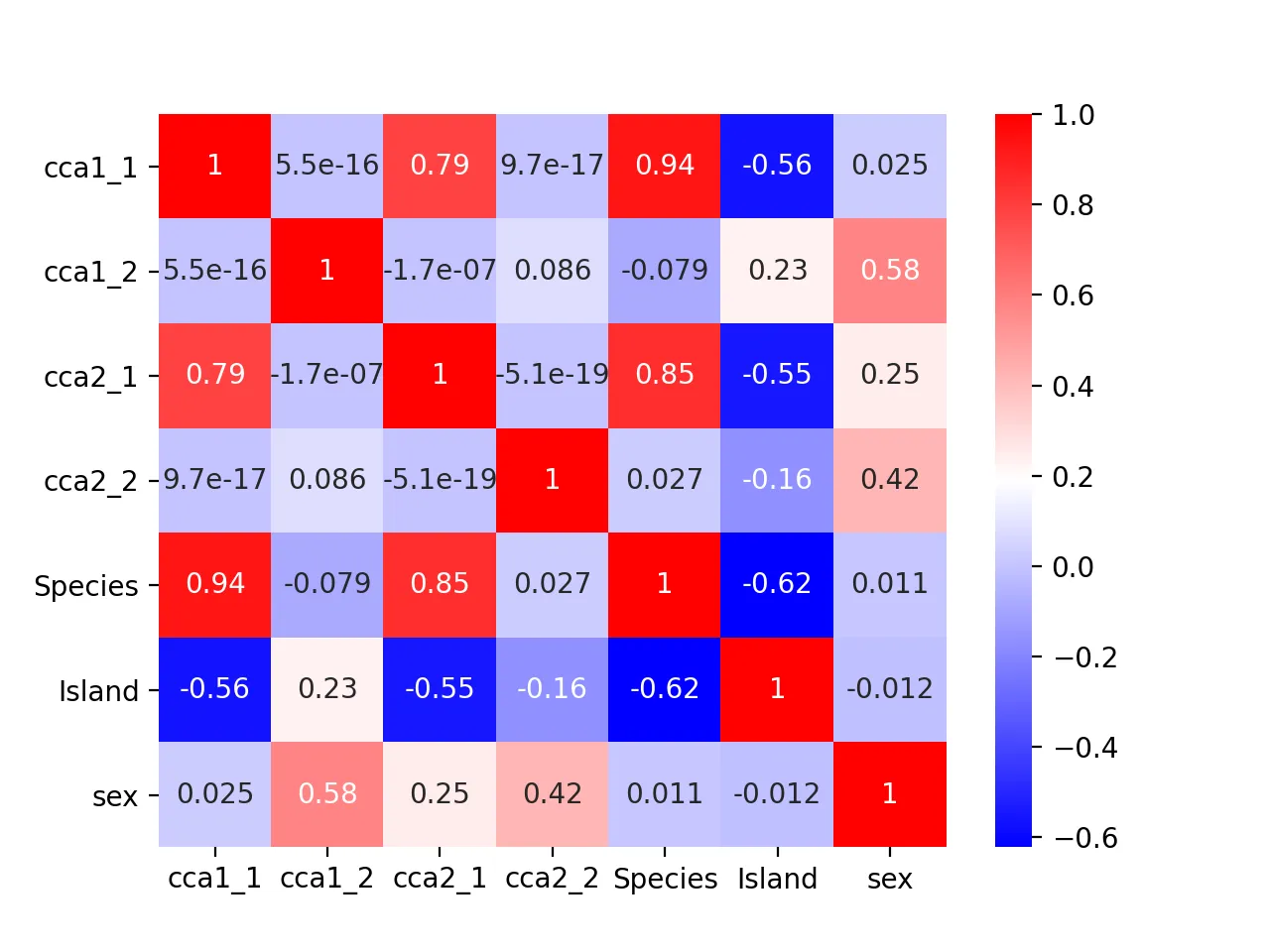
可以看到sex性别与cca2_2相关性最大,为0.42,推测其隐藏信息是性别
sns.scatterplot(data=cca_res,x="CCA1_2",y="CCA2_2",hue="sex",s=10)
plt.title(f'Second column corr = {np.corrcoef(cca_res.CCA1_2,cca_res.CCA2_2)[0, 1]:.2f}')
plt.savefig("cca_sex.png",dpi=200)
plt.close()
图为:
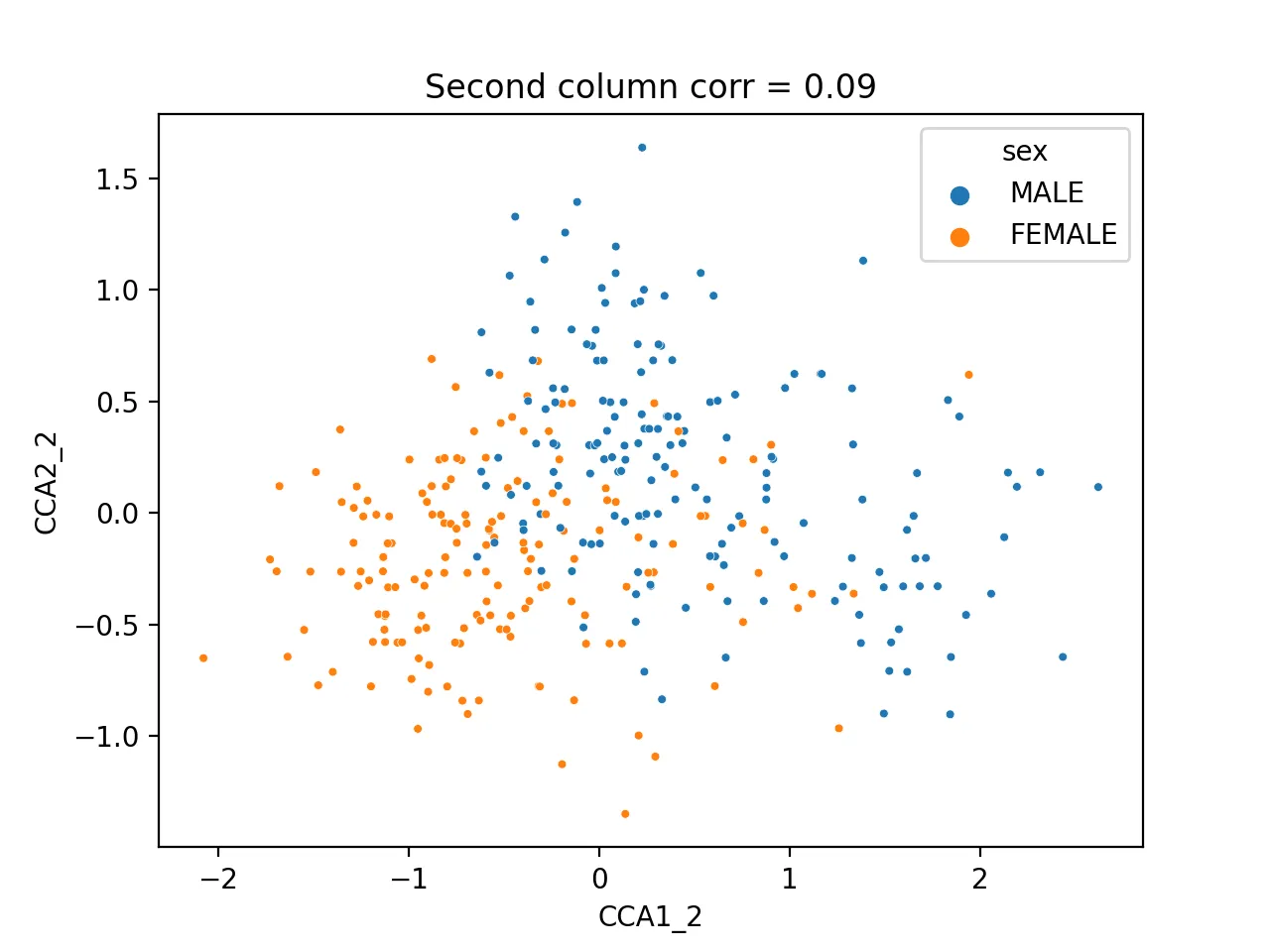 不同样本可以明显区分开来,
不同样本可以明显区分开来,
CCA在高维数据中可以很好的进行多组类型数据的处理,在企鹅数据中表现很好,在生物学上多组学数据的联合分析也是基础的处理方法,后续继续介绍其他的多组学处理方法.
Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK