
3

树空间的多维缩放(multidimensional scaling,MDS)可视化物种树和基因树的RF距离
source link: https://yanzhongsino.github.io/2023/03/13/bioinfo_phylogeny_MDS/
Go to the source link to view the article. You can view the picture content, updated content and better typesetting reading experience. If the link is broken, please click the button below to view the snapshot at that time.
1. 树空间的多维缩放(multidimensional scaling,MDS)可视化物种树和基因树的RF距离
- 当系统发育拓扑冲突时,需要评估基因树与物种树的不一致程度,可使用树空间的多维缩放(multidimensional scaling,MDS)来对树的RF距离进行降维,并在指定维度可视化基因树与物种树之间的RF距离。
- 先使用R包ape 5.0计算树之间的成对Robinson-Foulds(RF)距离,然后对RF距离进行多维缩放(multidimensional scaling,MDS),最后绘制指定维度下的RF距离示意图。
- MDS的二维图如下所示,图中的物种树,包括不同数据和不同软件的物种树(包括ASTRAL_tree,SCGs_ML,SNPs_ML,Plastome_ML树)用其他颜色标示,每个蓝色的点代表一棵基因树,颜色深浅代表支持率平均值的大小。
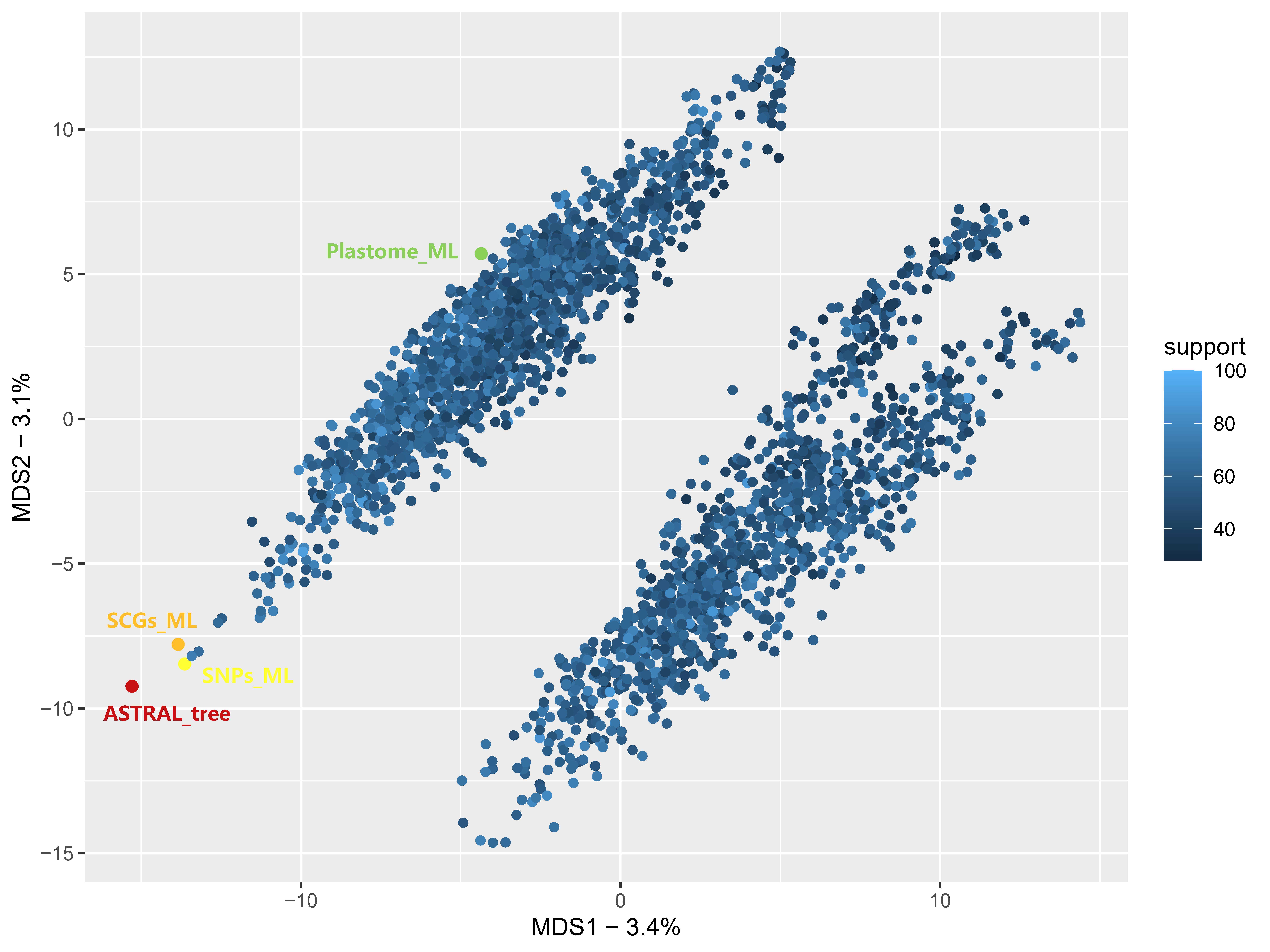
图 1. MDS二维图示例
2. 分析过程
- species_genes.tre:物种树和基因树合并的树文件,可以合并多棵物种树(比如用不同算法和数据构建的树)。
- 用R包ape计算基因树成对间的Robinson-Foulds(RF)距离
library(ape) # 载入ape包
trees<-read.tree("species_genes.tre") # 读取树
trees_dist<-dist.topo(trees, method = "PH85") # 计算RF,trees_dist的数据类别是dist
trees_matric<-as.matrix(trees_dist) # 转化dist到矩阵matrix
write.table (trees_matric, file ="RF_pairs.tsv", sep ="\t") # 保存矩阵数据到RF_pairs.tsv文件
- RF_pairs.tsv文件是行和列都是tree1,tree2,…,treen等依次命名的树之间的RF距离矩阵。
- MDS多维缩放
- 经典MDS法
mds <- cmdscale(trees_dist, eig=TRUE, k=2) # k代表维度
mds.var.per <- round(mds$eig/sum(mds$eig)*100,1)
mds.values <- mds$points
mds.data <- data.frame(Sample=rownames(mds.values), X=mds.values[,1], Y=mds.values[,2])
write.table(mds.data,file="mds.tsv",sep="\t") # 把MDS的二维值保存到mds.tsv
- mds.tsv文件示例。每棵树都有一行数据,这里是二维的,包含MDS1和MDS2两个值。用tree1,tree2,…,treen等依次命名,与species_genes.tre树文件中树的顺序一致。如果合并了物种树和基因树,可以依据顺序在mds.tsv中找到物种树对应的行。
"Sample" "MDS1" "MDS2"
"tree1" "tree1" -15.2853071675022 -9.23665453167777
"tree2" "tree2" -13.8416686613703 -7.78853307096205
- nonmetric MDS法
library(MASS)
mds <- isoMDS(trees_dist, k=2) # k代表维度
library(ggplot2)
ggplot(data=mds.data, aes(x=X, y=Y, label=Sample))+
geom_text() +
xlab(paste("MDS1 - ", mds.var.per[1], "%", sep=""))+
ylab(paste("MDS2 - ", mds.var.per[2], "%", sep=""))+
theme_bw()+
ggtitle("MDS plot using Euclidean distance")
mds <- cmdscale(trees_dist, eig=TRUE, k=2) # k代表维度
x <- mds$points[,1]
y <- mds$points[,2]
plot(x, y, xlab="Coordinate 1", ylab="Coordinate 2", main="Metric MDS", type="n")
text(mds$points, labels = rownames(trees_matric), cex = 0.6)
结果图保存成pdf后可以在pdf编辑器里把物种树对应的点用更大尺寸和其他颜色标示出来。
- 还可以把每个基因树和物种树的支持率的平均值计算出来之后,用支持率给每棵树的MSD点上色,从而查看是否是离物种树越近的基因树的平均支持率越高。
- 也可以用基因树的平均支持率和基因树离物种树的RF距离画散点图,并做线性回归分析判断相关性。
3. references
- 欢迎关注微信公众号:生信技工
- 公众号主要分享生信分析、生信软件、基因组学、转录组学、植物进化、生物学概念等相关内容,包括生物信息学工具的基本原理、操作步骤和学习心得。

Recommend
About Joyk
Aggregate valuable and interesting links.
Joyk means Joy of geeK